Dezember 2017
Ergebnisse 4. Teil
Die ribosomale DNA (rDNA) als geographischer Marker bei Cyclamen purpurascens hat sich inzwischen bewährt. Nochmal kurz zur rDNA:
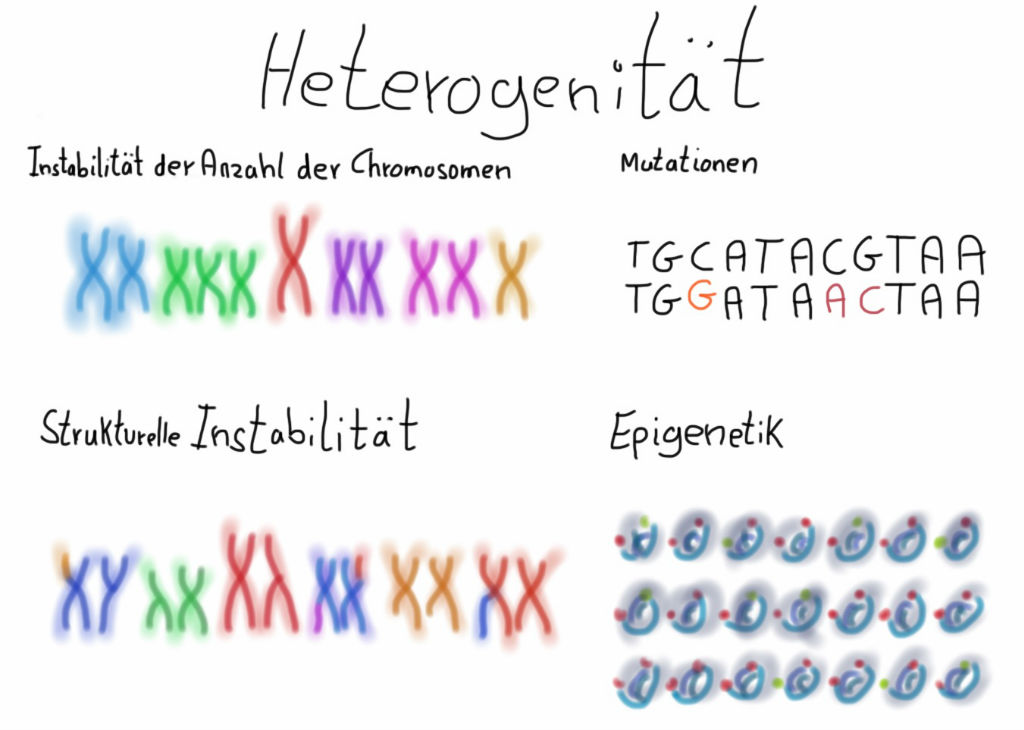
Die rDNA kodiert die Gene für ribosomale RNA, also einen Großteil der Ribosomen. In eukaryotischen Zellen gibt es rDNA nicht nur im Zellkern, sondern auch in den Mitochondrien, und bei Pflanzen zusätzlich in den Plastiden. Dazu kommt, dass es ein Repeat ist, sich also ziemlich oft wiederholt. Dies alles verursacht bei der Sequenzierung Probleme, auf die ich hier nicht näher eingehen will.
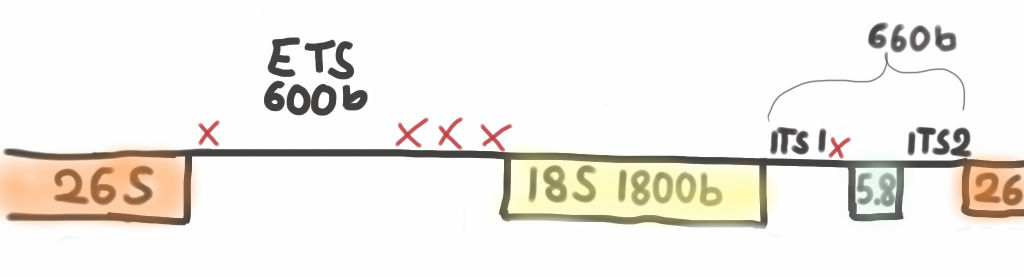
Gerade die Spacer zwischen den einzelnen Transkriptionseinheiten sind für die Unterscheidung sehr interessant. Dies sind ETS, ITS1und ITS2

rDNA-Repeat der ribosomalen Untereinheiten. Der external transcribed spacer (ETS) und die internal transcribed spacer (ITS1 + ITS2) befinden sich zwischen den Transkriptionseinheiten.
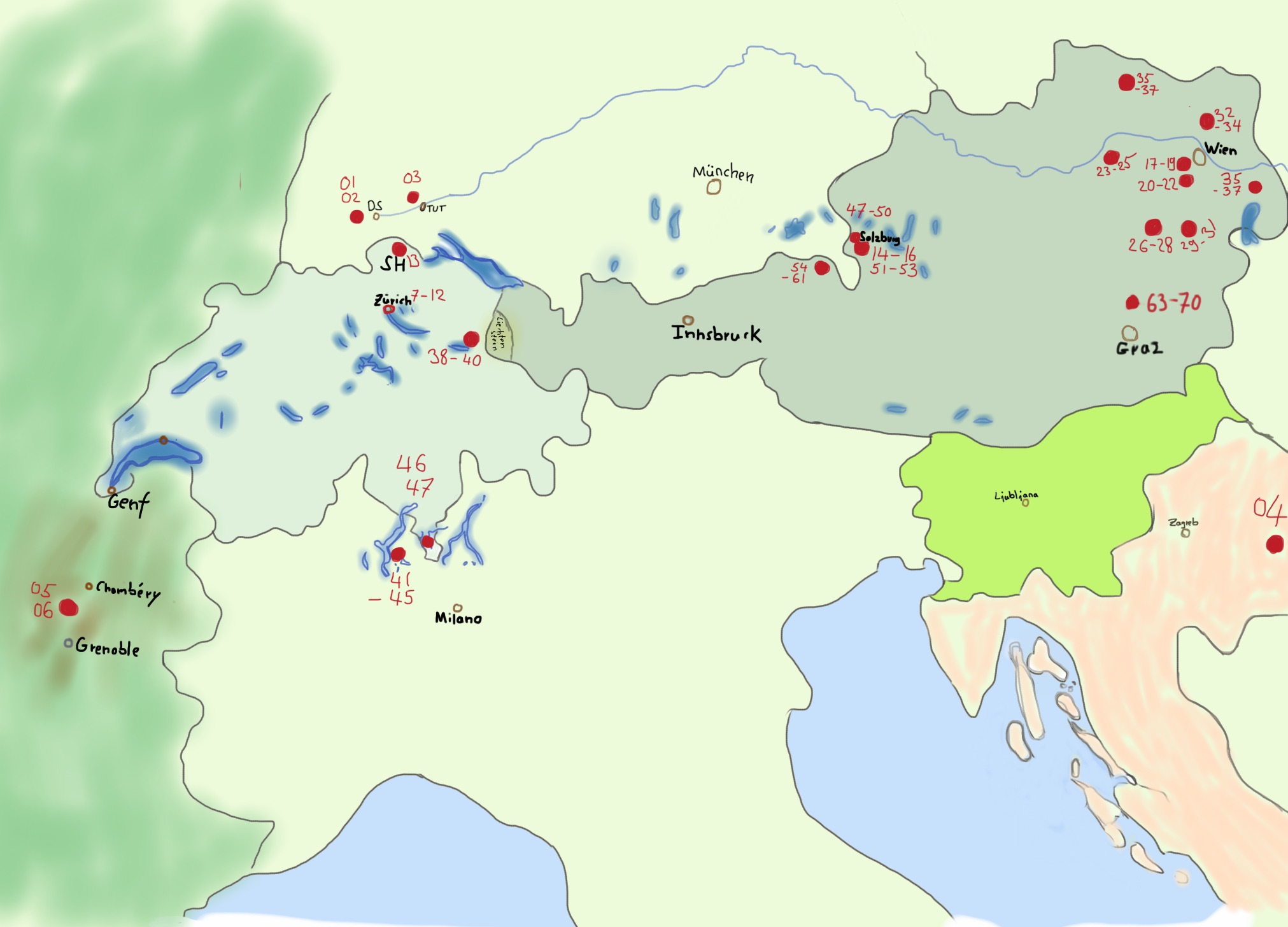
Vor allem mit Hilfe des Forum Flora Austria konnte ich viele verschiedene Blätter von Cyclamen purpurascens aus Österreich bekommen und werde wohl auch noch viele bekommen. An dieser Stelle nochmals vielen Dank an alle Beteiligten! Ihr habt mich einen großen Schritt weiter gebracht.
Durch einen kleinen Urlaub in Italien habe ich jetzt 70 Blätter in meiner Sammlung und auch schon einen großen Teil davon sequenziert.
Man kann zwei Sequenzfiles namens AB1 mit einem Programm namens SeqDoc vergleichen. Oben und unten sind die verschiedenen Sequenzen und in der Mitte die Gemeinsamkeiten (flacher Peak) und Unterschiede (großer Peak).
Ein Unterschied zwischen den Pflanzen aus Österreich und der Schweiz liegt vorne auf dem ETS. So ist nach den GGG bei den Pflanzen aus der Schweiz und Italien nur ein C, bei den Österreichischen Pflanzen zwei CC vor den vier AAAA. Hier die Peaks:
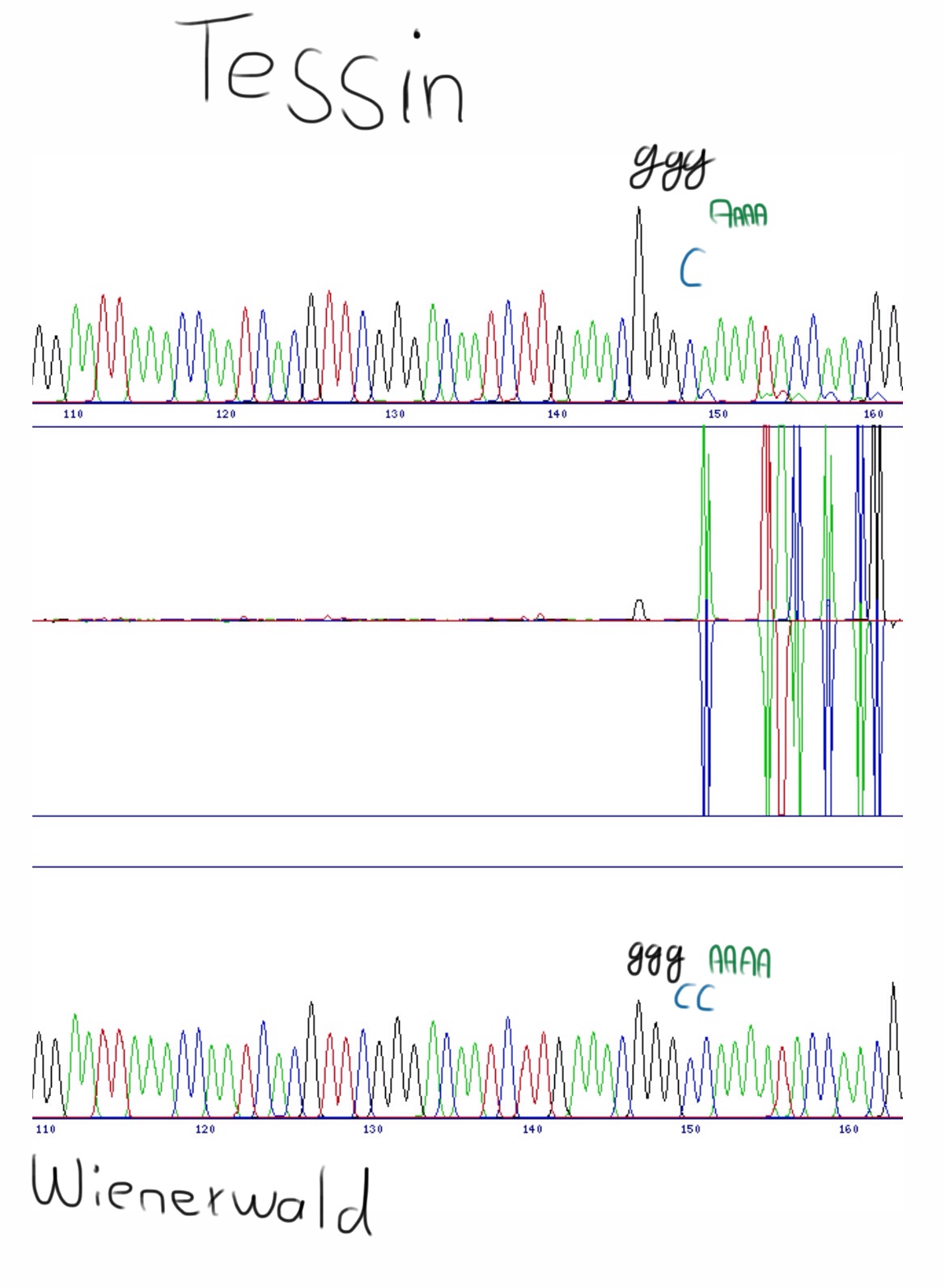
Vergleich von 50 Basen auf dem ETS zwischen einer Pflanze aus dem Tessin und einer aus Niederösterreich.
Ein anderer auffälliger Unterschied ist im ITS1. Hier ist ebenfalls bei den Österreichischen Pflanzen eine Base (G) mehr.

Chromatogramm mit zwei AB1 files von der Sequenzierung mit SeqDoc.
Am Anfang vom ETS kann man schon die verschiedenen Standorte unterscheiden. Die rDNA Sequenzen der verschiedenen Standorte wurde mit dem Programm Multi Sequence Alignment by CLUSTALW verglichen. Hier die Sequenz vom ETS vorne mit ausgewählten Pflanzen:

5′ Ende vom ETS mit 50 Basen von ausgewählten Standorten.
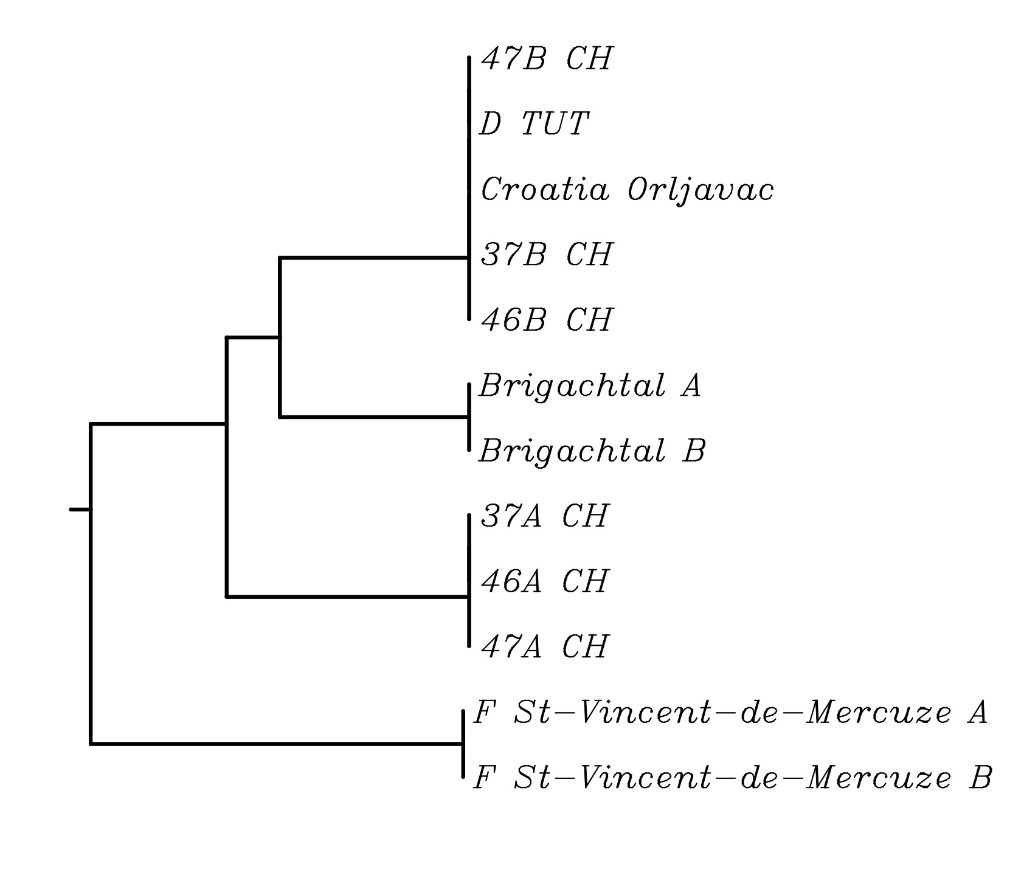
Mit dem dem Programm Multi Sequence Alignment by CLUSTALW kann man dann einen polygenetischen Baum mit den Sequenzen der rDNA erstellen.
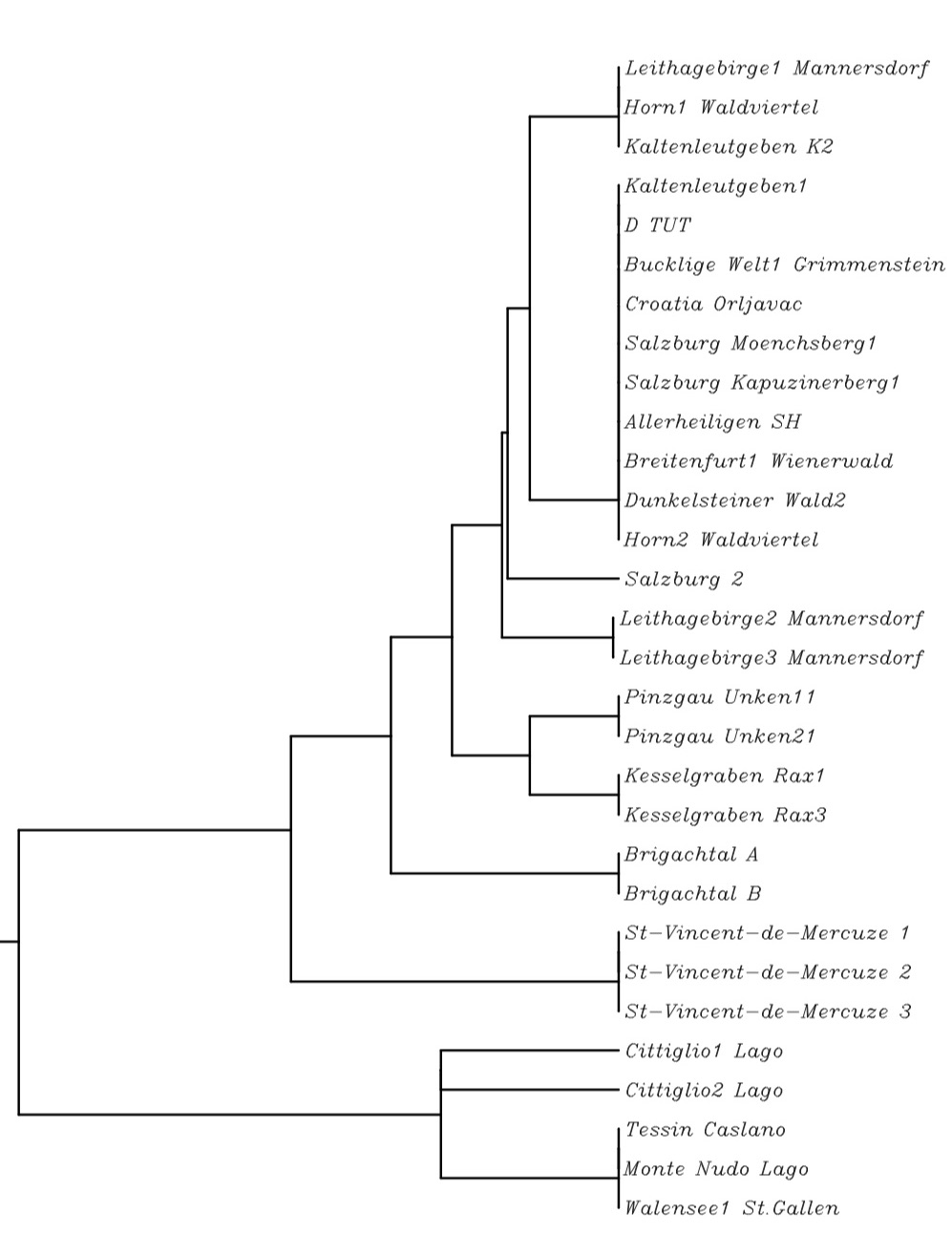
Polygenetischer Baum der rDNA ausgewählter Standorte von Cyclamen purpurascens.
Kommen wir zu dem Rätsel, warum die Pflanzen aus dem Klostergarten Allerheiligen in Schaffhausen, die Pflanze aus Orljavac in Kroatien und die Tuttlinger Pflanze aus der Nähe vom Kraftstein die selbe Sequenz wie die Pflanzen aus Österreich haben.
Die Pflanze aus Kroatien stammt aus der Nähe der Benediktioner Abtei des heiligen Mihovil.
Das Kloster Allerheiligen wurde ebenso von den Benediktinern gegründet und bei Tuttlingen befindet sich das Kloster Beuron mit der Erzabtei St. Martin.
Die Benediktiner legten schon früh Heilpflanzengärten an und brachten viele Pflanzen über die Alpen. Damals war es auch üblich, dass einmal im Jahr Bauern eingeladen wurden, um in den Gärten unterrichtet zu werden. Dieser Brauch war noch bis 1911 im Salzburgischen Kloster üblich. Salzburg war und ist mit der Erzabtei St. Peter ein Zentrum der Benediktiner. “Wolfgang Erdäpferl” wurden Cylamen purpurascens von den Einheimischen genannt und es wurde ihm eine besonders heilende Wirkung bei Schlangenbiß, Migräne, Kopfbeschwerden und als Zaubertrank nachgesagt. (Siehe Salzburgwiki).
Von wo aus genau -Salzburg oder Wien- die Benediktiner das Alpenveilchen ursprünglich verteilt haben, lässt sich nicht genau sagen. Es gibt sowohl im Salzburger Land, als auch in Niederösterreich Cyclamen mit der selben Sequenz der rDNA. Allerdings sind die Pflanzen um Wien einheitlich und die Pflanzen um Salzburg weisen zum Teil Variationen auf. Es läßt sich also spekulieren, dass um Salzburg das “Erdapferl”, das im großen Stiel als Heil und Fruchtbarkeits-Symbol für Pilger gesammelt und verkauft wurde, dadurch vielleicht etwas selten wurde. Womöglich wurde um Salzburg denn wieder mit Pflanzen aus den Klostergärten “aufgeforstet”.

Klostergarten Allerheiligen in Schaffhausen.
Cyclamen purpurascens war für die Benediktiner eine wichtige Heilpflanze die in den Klostergärten zu finden war und ist. Allem Anschein nach wurde das Alpenveilchen von den Benediktinern aus Österreich über die Ränder der Alpen hin verteilt. Wenn die Bedingungen für die Pflanzen gut waren, kann man sie heute noch finden.
Allerdings stammt das Cyclamen purpurasecens im Brigachtal nicht aus Österreich.
Woher es stammt, wird die Zukunft zeigen. Es fehlen mir noch einige natürliche Standorte.
Text und Abbildungen dürfen unter den Bedingungen der Creative Commons Attribution Share-Alike license (CC-BY-SA) gerne ganz oder teilweise kopiert und zitiert werden.







 tt ohne Adern. Es sollten nicht mehr als 50 mg sein. Dieses Blatt wird zerkleinert. Man kann dazu einen herkömmlichen Mörser nehmen und mörsert am besten mit flüssigem Stickstoff. Wenn dies nicht vorhanden ist, geht auch solch ein blauer Plastikmöppel ohne Stickstoff. Nur sollte man sich dabei beeilen und möglichst im Puffer zerkleinern. Der blaue Plastikmöppel heißt “Micro Pestel”.
tt ohne Adern. Es sollten nicht mehr als 50 mg sein. Dieses Blatt wird zerkleinert. Man kann dazu einen herkömmlichen Mörser nehmen und mörsert am besten mit flüssigem Stickstoff. Wenn dies nicht vorhanden ist, geht auch solch ein blauer Plastikmöppel ohne Stickstoff. Nur sollte man sich dabei beeilen und möglichst im Puffer zerkleinern. Der blaue Plastikmöppel heißt “Micro Pestel”.